| ← |
«Секс на R» |
→ |
| Секс на R–09: Моделювання парадокса Мейнарда Сміта (Завдання I) | Секс на R–10: Моделювання циклічно змінюваного добору за Стертевантом та іншими Завдання II |
Секс на R–11: Чому статей саме дві? (Опис проблеми III) |
Моделювання циклічно змінюваного добору за Стертевантом та іншими
8.1 Задача моделі
Один з можливих механізмів, що могли б забезпечувати підтримку статевого відтворення в популяціях, запропонував Альфред Стертевант. Модель Стертеванта стосувалася епістатичного добору, якій спочатку підтримував одні генні сполучення, а потім — інші. Назва епістатичного добору за Стертевантом пов'язана з явищем епістазу — взаємодії генів. Вільям Гамільтон та Олексій Кондрашов зрозуміли, що цей механізм має працювати і у разі змін характеру добору на користь різних сполучень аллелів за тим самим геном. Характер добору, який розглядали Гамільтон та Кондрашов можна назвати циклічно змінюваним.
Логіка цього процесу показана у таблиці.
Таблиця 8.1.1 Циклічно змінюваний добір, що сприяє то гетерозиготам, то гомозиготам
|
Покоління |
Характер добору |
AA |
Aa |
aa |
|
Генотипи |
||||
|
1 |
непарні: знищуються гомозиготи |
|
Aa |
|
|
2 |
парні: знищуються гетерозиготи |
AA |
|
aa |
|
3 |
непарні: знищуються гомозиготи |
|
Aa |
|
|
4 |
парні: знищуються гетерозиготи |
AA |
|
aa |
|
… |
… |
|
|
|
Цю ідею варто перевірити за допомогою імітаційної моделі. На початку такого дослідження можна впевнитися, що описаний механізм дійсно сприятиме сексуальному відтворенню роздільностатевих організмів та заважатиме партеногенетикам. Цікаво встановити, який добір компенсує «подвійну вартість статі». Крім того, цікаво впевнитися, чи вірним є припущення, що найуспішнішими у таких умовах будуть перехреснозапліднювані гермафродити.
8.2 Опис концептуальної моделі «Sturtevant etc»
Розглянемо найпростіший випадок циклічно змінюваного добору, що впливає на популяцію, до складу якої можуть входити самиці, самці, перехреснозапліднювані гермафродити та партеногенетики. Чисельність цієї популяції має обмеження K, кількість потомків кожної самиці, гермафродита та партеногенетика становить r.
Будемо спочатку вважати, що самиці схрещуються лише з самцями, а гермафродити — з гермафродитами (хоча можна розглянути й можливість схрещування самиць з гермафродитами та самців з гермафродитами). У такому разі кількість потомків є наступною:
maOFe(×Ma) = r/2;
hpOHp(×Hp) = r;
pgOPg = r.
Ця кількість потомків вказана у розрахунку на одну самицю, гермафродита або партеногенетика. При схрещуванні самиці та самця утворюється один виводок сумарною чисельністю r (який продукує самиця), а при схрещуванні гермафродитів — два, кожен по r, який продукують обидва партнери.
Кожна особина має певний генотип та спосіб відтворення. Склад популяції будемо кодувати набором чисел у векторі. Можливі варіанти кодів показані в таблиці.
Таблиця 8.2.1 Числові позначення способів відтворення та генотипів особин, що будуть використані у моделі
|
Кодування генотипів та характеру відтворення особин у моделі |
Генотипи |
|||||
|
Гомозигота за домінантним алелем |
Гетерозигота |
Гомозигота за рецесивним алелем |
||||
|
AA |
Aa |
aa |
||||
|
+0.11 |
+0.12 |
+0.22 |
||||
|
Тип відтворення |
Самиці |
Fe |
1+ |
1.11 |
1.12 |
1.22 |
|
Самці |
Ma |
2+ |
2.11 |
2.12 |
2.22 |
|
|
Гермафродити |
Hp |
3+ |
3.11 |
3.12 |
3.22 |
|
|
Партеногенетики |
Pg |
4+ |
4.11 |
4.12 |
4.22 |
|
Як зазначено раніше, ми будемо вважати, що в непарних (1, 3 тощо) поколіннях діє добір з силою s проти гомозигот, а в парних поколіннях (2, 4 тощо) — добір з цією ж самою силою проти гетерозигот. У розумінні сили (чи коефіцієнту) добору ми будемо виходити з визначення, що відповідає класичним зразкам:
«Селективну перевагу одного алеля перед альтернативним алелем (або алелями) можна виразити у відсотках або у вигляді коефіцієнту добору (s), величина якого змінюється в діапазоні від 0 до 1. Кількісне значення коефіцієнта добору виводиться із відносних темпів відтворення альтернативних алелей. Припустимо, що в якійсь великій популяції а — алель, що має перевагу, а А — алель, якому добір не сприяє. У цій популяції на кожні 100 алелей а, переданих наступному поколінню, буде передаватися також кілька алелей А (від 100 до 0). Коефіцієнт добору є функцією цього відношення. Величину s можна визначити за формулою
s = 1 – (Швидкість відтворення алеля, якому не сприяє добір/Швидкість відтворення алеля, якому сприяє добір)»
Грант В. Эволюционный процесс, 2008
Наприклад, добір з силою s проти гомозигот означає, що в цьому поколінні ймовірність виживання (не пов’язаного зі скороченням чисельності) гетерозигот дорівнює 1 (а ймовірність смертності — 0), а ймовірність виживання гомозигот 1–s (а ймовірність смертності — s). Фактично, в нашій моделі ми будемо мати справу з двома видами смертності. Перша — селективна (пов’язана з дією добору). Друга — неселективна; вона спостерігатиметься у разі, якщо після селективної смертності чисельність популяції N буде перевищувати K, тобто у разі (N>K). У такому разі ймовірність загибелі будь-якої особини, яка збереглася після селективної смертності, має бути однаковою і дорівнювати (N–K)/N. Ймовірність виживання, відповідно, дорівнюватиме 1–(N–K)/N.
Передбачимо в життєвому циклі модельної популяції такі етапи:
↓ розмноження
βP — бета-сукупність — склад після розмноження; кількість батьків + кількість потомків;
↓ селективна смертність
γP — гамма-сукупність — результат добору серед потомків (характер добору буде змінюватися у різних циклах);
↓ неселективна смертність
ωP — омега-сукупність — остаточний склад особин на кінець циклу (що визначає альфа-сукупність на наступному етапі циклу).
Результатом роботи моделі I типу буде динаміка особин з різними типами відтворення, моделі II типу — розподіл ймовірності результатів, а моделі III типу — залежність наймовірніших результатів від значення початкових параметрів (насамперед, s).
8.3 Покрокова побудова моделі «Sturtevant etc» в R: модель I типу
Використаємо для побудови моделі типову структуру, що міститься у наступному шаблоні.
# Models name — назва моделі
# I. ENTRANCE — ВХІД:
# I.1. Initial script commands — початкові команди скрипту
# I.2. Initial state of the system — початковий стан модельованої системи
# I.3. Parameters — параметри
# I.4. Experimental conditions — умови експерименту з моделлю
# I.5. The changeable parameters combinations — комбінації змінюваних параметрів
# II TRANSFORMATIONS SYSTEM CREATION — СТВОРЕННЯ СИСТЕМИ ПЕРЕТВОРЕНЬ:
# II.1. Users function setting — створення користувацьких функцій
# II.2. Parameters combinations mechanism — механізм комбінації змінюваних параметрів
# II.3. Objects creation — створення об'єктів
# II.4. Calculation rules data — Дані з правилами перерахунків
# III. CALCULATIONS — РОЗРАХУНКИ:
# III.1. Higher level cycles running — запуск циклів вищого рівня
# III.2. Initial composition creation — утворення початкового складу
# III.3. Main work cycle — основний робочий цикл
# III.4. Ending of higher level cycles — завершення циклів вищого рівня
# IV. FINISHING — ЗАВЕРШЕННЯ:
# IV.1. Creating objects for results integration — створення об'єктів, що інтегрують результати
# IV.2. Results saving — збереження об'єктів з результатами
# IV.3. Results viewing — перегляд підсумків
# IV.4. Results visualization — візуалізація підсумківЗрозуміло, що далеко не в кожній моделі спостерігатиметься весь набір перелічених елементів. З іншого боку, у розділі «III.3. Main work cycle — основний робочий цикл» може виникати необхідність у створенні підрозділів.
Починаємо. Реалізуємо такий варіант: створюється популяція з певною чисельністю різних форм у її складі, а потім цим особинам випадково присвоюється генотип за геном A. Ймовірність різних генотипів визначатиметься вектором Genot.
Обрано рішення, за якого у матрицю Rez заносяться усі 12 форм особин, що є у модельній популяції (4 форми розмноження × 3 генотипи). Це — неекономне рішення, але наслідком його буде можливість отримати з матриці Rez усю суттєву інформацію.
# Sturtevant etc: ВПЛИВ ЧЕРГУВАННЯ ТИПІВ ДОБОРУ НА ХАРАКТЕР ВІДТВОРЕННЯ В ПОПУЛЯЦІЇ
# Модель I типа (однократна імітація)
# I. ENTRANCE — ВХІД:
# I.1. Initial script commands — початкові команди скрипту
# setwd("...") # Робоча директорія
rm(list = ls()) # Очищення раніше збережених об'єктів в Environment
set.seed = 12345 # Використання певного варіанту вибору випадкових чисел
# I.2. Initial state of the system — початковий стан модельованої системи
al_0_N_fe <- 8 # Початкова чисельність самиць
al_0_N_ma <- 8 # Початкова чисельність самців
al_0_N_hp <- 2 # Початкова чисельність перехреснозапліднюваних гермафродитів
al_0_N_pg <- 2 # Початкова чисельність партеногенетиків
Genot <- c(0.11, 0.12, 0.12, 0.22) # Початковий розподіл генотипів: 11 = AA, 12 = Aa, 22 = aa
# I.3. Parameters — параметри
K <- 20 # Обмеження ємності середовища
r <- 6 # Плодючість (розмір виводка самиці, гермафродита або партеногенетика)
fe_O_FeMa <- r/2 # Кількість самиць у потомстві самиці (що була запліднена самцем)
ma_O_FeMa <- r/2 # Кількість самців у потомстві самиці (що була запліднена самцем)
hp_O_HpHp <- r # Кількість гермафродитів у потомстві гермафродита (що спарувався з іншим гермафродитом)
pg_O_Pg <- r # Кількість партеногенетиків у потомстві партеногенетика (якому не треба з кимось паруватися)
s <- 0.7 # Сила циклічно змінюваного негативного добору
# I.4. Experimental conditions — умови експерименту з моделлю
cycles <- 10 # Кількість робочих циклів моделі
# II TRANSFORMATIONS SYSTEM CREATION — СТВОРЕННЯ СИСТЕМИ ПЕРЕТВОРЕНЬ:
# II.3. Objects creation — створення об’єктів
rws <- c("Females_AA", "Females_Aa", "Females_aa", "Males_AA", "Males_Aa", "Males_aa", "Hermaph_AA", "Hermaph_Aa", "Hermaph_aa", "Parthen_AA", "Parthen_Aa", "Parthen_aa") # Рядки для матриці з підсумками
cls <- c(0:cycles) # Позначення стовпців для матриці з підсумками
Rez <- matrix(NA, nrow = length(rws), ncol = length(cls), dimnames = list(rws, cls)) # Створення матриці для запису підсумків
Al_Pop <- rep(NA, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg) # Створення вектору для початкової чисельності
# III. CALCULATIONS — РОЗРАХУНКИ:
# III.2. Initial composition creation — утворення початкового складу
if(al_0_N_fe>0) Al_Pop[1:al_0_N_fe] <- 1 # У файл для альфа-чисельності переносяться самиці...
if(al_0_N_ma>0) Al_Pop[(al_0_N_fe+1):(al_0_N_fe+al_0_N_ma)] <- 2 # ... самці...
if(al_0_N_hp>0) Al_Pop[(al_0_N_fe+al_0_N_ma+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp)] <- 3 # ... гермафродити...
if(al_0_N_pg>0) Al_Pop[(al_0_N_fe+al_0_N_ma+al_0_N_hp+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp+al_0_N_pg)] <- 4 # ... партеногенетики
Al_Pop <- Al_Pop + sample(Genot, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg, replace=T) # Випадково визначаються генотипи усіх особин
Al_Pop
## [1] 1.12 1.12 1.12 1.22 1.12 1.12 1.11 1.12 2.12 2.12 2.11 2.12 2.12 2.12 2.11
## [16] 2.12 3.11 3.22 4.12 4.12Склад модельної популяції відповідає задуму…
Перенесемо альфа-склад у матрицю Rez. У подальших розрахунках будемо використовувати окремі значення з цієї матриці. Оскільки основний робочий цикл поки що не дописаний до кінця, заблокуємо його початок «ґраткою» і вкажемо, що поки що ми конструюємо перший цикл.
# III.3. Main work cycle — основний робочий цикл
# III.3.a. Початок основного робочого циклу моделі
# for (t in 1:cycles) {
t <- 1 # Тимчасова умова, яку уведено, щоб відтестувати елементи робочого циклу моделі до того, як його ввімкнено
if(t>1) Al_Pop <- na.omit(Om_Pop) # В циклах крім першого Al_Pop - це Om_Pop попереднього циклу без NA
# III.3.b. Створюються та переносяться у матрицю результатів позначення чисельності початкових груп
Rez["Females_AA", t] <- length(which(Al_Pop==1.11))
Rez["Females_Aa", t] <- length(which(Al_Pop==1.12))
Rez["Females_aa", t] <- length(which(Al_Pop==1.22))
Rez["Males_AA", t] <- length(which(Al_Pop==2.11))
Rez["Males_Aa", t] <- length(which(Al_Pop==2.12))
Rez["Males_aa", t] <- length(which(Al_Pop==2.22))
Rez["Hermaph_AA", t] <- length(which(Al_Pop==3.11))
Rez["Hermaph_Aa", t] <- length(which(Al_Pop==3.12))
Rez["Hermaph_aa", t] <- length(which(Al_Pop==3.22))
Rez["Parthen_AA", t] <- length(which(Al_Pop==4.11))
Rez["Parthen_Aa", t] <- length(which(Al_Pop==4.12))
Rez["Parthen_aa", t] <- length(which(Al_Pop==4.22))
Rez
## 0 1 2 3 4 5 6 7 8 9 10
## Females_AA 0 NA NA NA NA NA NA NA NA NA NA
## Females_Aa 6 NA NA NA NA NA NA NA NA NA NA
## Females_aa 2 NA NA NA NA NA NA NA NA NA NA
## Males_AA 1 NA NA NA NA NA NA NA NA NA NA
## Males_Aa 4 NA NA NA NA NA NA NA NA NA NA
## Males_aa 3 NA NA NA NA NA NA NA NA NA NA
## Hermaph_AA 0 NA NA NA NA NA NA NA NA NA NA
## Hermaph_Aa 0 NA NA NA NA NA NA NA NA NA NA
## Hermaph_aa 2 NA NA NA NA NA NA NA NA NA NA
## Parthen_AA 1 NA NA NA NA NA NA NA NA NA NA
## Parthen_Aa 1 NA NA NA NA NA NA NA NA NA NA
## Parthen_aa 0 NA NA NA NA NA NA NA NA NA NAНа жаль, розрахунок чисельності потомства з різними генотипами потребує врахування чисельності усіх можливих схрещувань. Спочатку треба розібратися, як їх розраховувати…
Ми маємо три генотипи самиць (їх чисельності позначені як Rez[“Females_AA”, t], Rez[“Females_Aa”, t] та Rez[“Females_aa”, t]) і три генотипи самців (їх чисельності позначені як Rez[“Males_AA”, t], Rez[“Males_Aa”, t] та Rez[“Males_aa”, t]). Можливі таки варіанти схрещування:
Females_AA × Males_AA → Females_AA : Males_AA
Females_AA × Males_Aa → Females_AA : Males_AA : Females_Aa : Males_Aa
Females_Aa × Males_AA → Females_AA : Males_AA : Females_Aa : Males_Aa
Females_AA × Males_aa → Females_Aa : Males_Aa
Females_aa × Males_AA → Females_Aa : Males_Aa
Females_Aa × Males_Aa → Females_AA : Males_AA : 2 Females_Aa : 2 Males_Aa : Females_aa : Males_aa
Females_Aa × Males_aa → Females_Aa : Males_Aa : Females_aa : Males_aa
Females_aa × Males_Aa → Females_Aa : Males_Aa : Females_aa : Males_aa
Females_aa × Males_aa → Females_aa : Males_aa
Скільки відбувається схрещувань, наприклад, Females_AA × Males_AA? Щоб розрахувати їх чисельність, корисно заздалегідь розрахувати загальну чисельність самців; ця кількість позначена як be_N_Ma. У такому разі
При таких розрахунках слід підстрахуватися від поділу на 0 у випадку, коли в модельній популяції не лишається самців. Якщо самців нема, бета-чисельність дорівнює альфа чисельності; якщо самці наявні, слід вирахувати кількість потомків від усіх можливих варіантів схрещування. Звісно, що кількість потомства від схрещувань залежить, крім іншого, від плодючості, яка задана в початкових параметрах.
fe_O_FeMa <- r/2 # Кількість самиць у потомстві самиці (що була запліднена самцем)
ma_O_FeMa <- r/2 # Кількість самців у потомстві самиці (що була запліднена самцем)А які можливі схрещування між гермафродитами?
Hermaph_AA × Hermaph_AA → Hermaph_AA
Hermaph_AA × Hermaph_Aa → Hermaph_AA : Hermaph_Aa
Hermaph_AA × Hermaph_Aa → Hermaph_Aa
Hermaph_Aa × Hermaph_Aa → Hermaph_AA : 2 Hermaph_Aa : Hermaph_aa
Hermaph_Aa × Hermaph_aa → Hermaph_Aa : Hermaph_aa
Hermaph_aa × Hermaph_aa → Hermaph_aa
Позначимо загальну кількість гермафродитів як be_N_Hp. У такому разі
Pairs_Herms_AA_AA <- Rez[“Hermaph_AA”, t]*Rez[“Hermaph_AA”, t]-1)/be_N_Hp.
Щоб вирахувати кількість партнерів Hermaph_AA для такої самої особини, ми маємо відняти одиницю від чисельності таких гермафродитів, адже ця особина не може бути партнером сама собі. Формули, що подібні до наведеної, можна лише у разі, якщо чисельність групи гермафродитів, яка розглядається, перевищує 0 (щоб не отримати від’ємні чисельності). Тому спочатку нам доведеться розрахувати кількість різних пар гермафродитів, а вже потім розрахувати чисельність потомків.
Як і у випадку роздільностатевих організмів, ми маємо передбачити умову, що спрощує розрахунки у разі, якщо чисельність гермафродитів знижується до критичного рівня. Якщо кількість гермафродитів менша за 2, їх відтворення зупиняється.
З врахуванням цього, ми можемо написати такий код.
# III.3.d. Розрахунок бета-чисельності (альфа-чисельність + потомство), з ймовірнісним округленням
be_N_Ma <- Rez["Males_AA", t] + Rez["Males_Aa", t] + Rez["Males_aa", t] # Загальна кількість самців
if(be_N_Ma==0) { # Якщо самців нема, бета-чисельність дорівнює альфа-чисельності
be_N_Fe_AA <- Rez["Females_AA", t]
be_N_Fe_Aa <- Rez["Females_Aa", t]
be_N_Fe_aa <- Rez["Females_aa", t]
be_N_Ma_AA <- Rez["Males_AA", t]
be_N_Ma_Aa <- Rez["Males_Aa", t]
be_N_Ma_aa <- Rez["Males_aa", t]
} else { # Якщо самці є, чисельність кожної форми розраховується залежно від схрещувань, де вона може утворюватися
be_N_Fe_AA <- Rez["Females_AA", t] + floor(fe_O_FeMa * # Альфа чисельність + ймовірнісне округлення добутку плодючості та кількості схрещувань, де утворюється потрібний генотип
(Rez["Females_AA", t] * Rez["Males_AA", t] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Fe_Aa <- Rez["Females_Aa", t] + floor(fe_O_FeMa *
(Rez["Females_AA", t] * Rez["Males_aa", t] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t] * Rez["Males_AA", t] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Fe_aa <- Rez["Females_aa", t] + floor(ma_O_FeMa *
(Rez["Females_aa", t] * Rez["Males_aa", t] / be_N_Ma + # aa × aa ⟶ aa
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # aa × Aa ⟶ Aa : aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_AA <- Rez["Males_AA", t] + floor(ma_O_FeMa *
(Rez["Females_AA", t] * Rez["Males_AA", t] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_Aa <- Rez["Males_Aa", t] + floor(ma_O_FeMa *
(Rez["Females_AA", t] * Rez["Males_aa", t] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t] * Rez["Males_AA", t] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Ma_aa <- Rez["Males_aa", t] + floor(ma_O_FeMa *
(Rez["Females_aa", t] * Rez["Males_aa", t] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
} # Закриття дужок підрахунку самиць та самців після else
be_N_Hp <- Rez["Hermaph_AA", t] + Rez["Hermaph_Aa", t] + Rez["Hermaph_aa", t] # Загальна кількість гермафродитів
if(be_N_Hp<2) { # Якщо гермафродитів менше за два, бета-чисельність дорівнює альфа-чисельності
be_N_Hp_AA <- Rez["Hermaph_AA", t]
be_N_Hp_Aa <- Rez["Hermaph_Aa", t]
be_N_Hp_aa <- Rez["Hermaph_aa", t]
} else { # Розраховується (з окремими позначеннями) чисельність різних за складом пар гермафродитів
if(Rez["Hermaph_AA", t]>1) Pairs_Herms_AA_AA <- Rez["Hermaph_AA", t]*(Rez["Hermaph_AA", t]-1)/be_N_Hp else Pairs_Herms_AA_AA <- 0
if(Rez["Hermaph_Aa", t]>1) Pairs_Herms_Aa_Aa <- Rez["Hermaph_Aa", t]*(Rez["Hermaph_Aa", t]-1)/be_N_Hp else Pairs_Herms_Aa_Aa <- 0
if(Rez["Hermaph_aa", t]>1) Pairs_Herms_aa_aa <- Rez["Hermaph_aa", t]*(Rez["Hermaph_aa", t]-1)/be_N_Hp else Pairs_Herms_aa_aa <- 0
Pairs_Herms_AA_Aa <- Rez["Hermaph_AA", t]*Rez["Hermaph_Aa", t]/be_N_Hp
Pairs_Herms_AA_aa <- Rez["Hermaph_AA", t]*Rez["Hermaph_aa", t]/be_N_Hp
Pairs_Herms_Aa_aa <- Rez["Hermaph_Aa", t]*Rez["Hermaph_aa", t]/be_N_Hp
# А тепер - бета-чисельність різних генотипів залежно від кількості можливих схрещувань
be_N_Hp_AA <- Rez["Hermaph_AA", t] + floor(hp_O_HpHp *
(Pairs_Herms_AA_AA*2 + # AA × AA ⟶ AA
Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_Aa_Aa/2) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Hp_Aa <- Rez["Hermaph_Aa", t] + floor(hp_O_HpHp *
(Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_AA_aa*2 + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_Aa_aa) + runif(1)) # Aa × aa ⟶ Aa : aa
be_N_Hp_aa <- Rez["Hermaph_aa", t] + floor(hp_O_HpHp *
(Pairs_Herms_Aa_aa + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa/2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_aa_aa*2) + runif(1)) # aa × aa ⟶ aa
} # Закриття дужок підрахунку гермафродитів після else
be_N_Pg_AA <- Rez["Parthen_AA", t] + floor(pg_O_Pg * Rez["Parthen_AA", t] + runif(1)) # ...і партеногенетики
be_N_Pg_Aa <- Rez["Parthen_Aa", t] + floor(pg_O_Pg * Rez["Parthen_Aa", t] + runif(1))
be_N_Pg_aa <- Rez["Parthen_aa", t] + floor(pg_O_Pg * Rez["Parthen_aa", t] + runif(1))
be_N_Fe_AA
## [1] 3
be_N_Fe_Aa
## [1] 17
be_N_Fe_aa
## [1] 11
be_N_Ma_AA
## [1] 4
be_N_Ma_Aa
## [1] 16
be_N_Ma_aa
## [1] 12
be_N_Hp_AA
## [1] 0
be_N_Hp_Aa
## [1] 0
be_N_Hp_aa
## [1] 14
be_N_Pg_AA
## [1] 7
be_N_Pg_Aa
## [1] 7
be_N_Pg_aa
## [1] 0Усі параметри бета-складу модельної популяції розраховані. Тепер слід імітувати добір. Використаємо позначення для розрахунку залишку від поділу: %%. Для парних t буде реалізована умова t%%2==0, а для непарних — t%%2==1. Залежно від цього буде розраховуватися частка гомо- або гетерозигот, що увійдуть у гама-склад модельної популяції.
# III.3.e. Визначення добору для гомозигот та гетерозигот: залишок від поділу визначає, який йде добір
if(t%%2==1) { # Якщо покоління непарне, то...
v_homozygotes <- 1-s # ...чисельність гомозигот скорочується, ...
v_heterozygotes <- 1 # ...а чисельність гетерозигот лишається постійною
} else { # Якщо покоління парне, то...
v_homozygotes <- 1 # ...чисельність гомозигот лишається постійною...
v_heterozygotes <- 1-s} # ...а чисельність гетерозигот скорочується
# III.3.f. Вплив добору на кожну з груп (розрахунок гама-чисельностей)
ga_N_Fe_AA <- floor(be_N_Fe_AA * v_homozygotes + runif(1)) # Додаток кожної бета-чисельності та...
ga_N_Fe_Aa <- floor(be_N_Fe_Aa * v_heterozygotes + runif(1)) # ...життездатності у даному поколінні...
ga_N_Fe_aa <- floor(be_N_Fe_aa * v_homozygotes + runif(1)) # ...зазнає ймовірнісного округлення:
ga_N_Ma_AA <- floor(be_N_Ma_AA * v_homozygotes + runif(1)) # ...це і є гама-чисельність
ga_N_Ma_Aa <- floor(be_N_Ma_Aa * v_heterozygotes + runif(1))
ga_N_Ma_aa <- floor(be_N_Ma_aa * v_homozygotes + runif(1))
ga_N_Hp_AA <- floor(be_N_Hp_AA * v_homozygotes + runif(1))
ga_N_Hp_Aa <- floor(be_N_Hp_Aa * v_heterozygotes + runif(1))
ga_N_Hp_aa <- floor(be_N_Hp_aa * v_homozygotes + runif(1))
ga_N_Pg_AA <- floor(be_N_Pg_AA * v_homozygotes + runif(1))
ga_N_Pg_Aa <- floor(be_N_Pg_Aa * v_heterozygotes + runif(1))
ga_N_Pg_aa <- floor(be_N_Pg_aa * v_homozygotes + runif(1))
# III.3.g. Створення вектору з гама-складом популяції
Ga_Pop <- c(rep(1.11, ga_N_Fe_AA), rep(1.12, ga_N_Fe_Aa), rep(1.22, ga_N_Fe_aa), rep(2.11, ga_N_Ma_AA), rep(2.12, ga_N_Ma_Aa), rep(2.22, ga_N_Ma_aa), rep(3.11, ga_N_Hp_AA), rep(3.12, ga_N_Hp_Aa), rep(3.22, ga_N_Hp_aa), rep(4.11, ga_N_Pg_AA), rep(4.12, ga_N_Pg_Aa), rep(4.22, ga_N_Pg_aa))
Ga_Pop
## [1] 1.11 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12 1.12
## [16] 1.12 1.12 1.12 1.22 1.22 1.22 2.11 2.12 2.12 2.12 2.12 2.12 2.12 2.12 2.12
## [31] 2.12 2.12 2.12 2.12 2.12 2.12 2.12 2.12 2.22 2.22 2.22 3.22 3.22 3.22 3.22
## [46] 3.22 4.11 4.11 4.12 4.12 4.12 4.12 4.12 4.12 4.12Гама-склад сформований. Після того, як ми імітували добір, нам треба забезпечити невибіркове скорочення чисельності модельної популяції до передбачуваного обмеження (K).
# III.3.h. Створення вектора долі (Fate) і вектора жереба (Lot)
if(length(Ga_Pop)>K) Fate <- rep(NA, length(Ga_Pop)) else Fate <- rep(NA, K) # Визначається довжина вектора долі
Fate[1:K] <- 1 # В векторі долі стільки одиниць, скільки має вижити особин, але вони розташовані впорядковано
Lot <- sample(Fate, length(Ga_Pop), replace = FALSE) # В векторі жереба одиниці та NA випадково переплутані
# III.3.i. Вектор жереба визначає остаточний склад в циклі
Om_Pop <- Ga_Pop * Lot # У векторі Om_Pop лишається K особин; загиблі особини - NA
Om_Pop
## [1] NA 1.12 1.12 1.12 NA NA NA NA 1.12 NA NA NA NA NA NA
## [16] 1.12 NA NA NA NA 1.22 NA 2.12 NA NA 2.12 NA NA NA NA
## [31] NA NA NA 2.12 2.12 NA 2.12 NA 2.22 2.22 2.22 NA NA NA NA
## [46] 3.22 4.11 NA 4.12 NA 4.12 NA 4.12 NA 4.12Працює: у складі модельної популяції з’явилися «дірки», ніби-то хтось пройшовся по популяції з кулемета, вибиваючи випадкових особин, але саме у такій кількості, яка є необхідною для встановлення необхідної чисельності популяції.
Лишилося небагато. Треба закінчити робочий цикл. Після цього логічно додати до матриці Rez склад модельної популяції, який був отриманий в кінці останнього циклу.
# III.3.j. Закінчення основного робочого циклу моделі і збереження результатів останнього циклу
# } # Тимчасово вімкнена фігурна дужка, що замикає робочий цикл
Rez["Females_AA", cycles+1] <- length(which(Om_Pop==1.11))
Rez["Females_Aa", cycles+1] <- length(which(Om_Pop==1.12))
Rez["Females_aa", cycles+1] <- length(which(Om_Pop==1.22))
Rez["Males_AA", cycles+1] <- length(which(Om_Pop==2.11))
Rez["Males_Aa", cycles+1] <- length(which(Om_Pop==2.12))
Rez["Males_aa", cycles+1] <- length(which(Om_Pop==2.22))
Rez["Hermaph_AA", cycles+1] <- length(which(Om_Pop==3.11))
Rez["Hermaph_Aa", cycles+1] <- length(which(Om_Pop==3.12))
Rez["Hermaph_aa", cycles+1] <- length(which(Om_Pop==3.22))
Rez["Parthen_AA", cycles+1] <- length(which(Om_Pop==4.11))
Rez["Parthen_Aa", cycles+1] <- length(which(Om_Pop==4.12))
Rez["Parthen_aa", cycles+1] <- length(which(Om_Pop==4.22))
# IV. FINISHING — ЗАВЕРШЕННЯ:
# IV.3. Results viewing — перегляд підсумків
Rez
## 0 1 2 3 4 5 6 7 8 9 10
## Females_AA 0 0 NA NA NA NA NA NA NA NA NA
## Females_Aa 6 5 NA NA NA NA NA NA NA NA NA
## Females_aa 2 1 NA NA NA NA NA NA NA NA NA
## Males_AA 1 0 NA NA NA NA NA NA NA NA NA
## Males_Aa 4 5 NA NA NA NA NA NA NA NA NA
## Males_aa 3 3 NA NA NA NA NA NA NA NA NA
## Hermaph_AA 0 0 NA NA NA NA NA NA NA NA NA
## Hermaph_Aa 0 0 NA NA NA NA NA NA NA NA NA
## Hermaph_aa 2 1 NA NA NA NA NA NA NA NA NA
## Parthen_AA 1 1 NA NA NA NA NA NA NA NA NA
## Parthen_Aa 1 4 NA NA NA NA NA NA NA NA NA
## Parthen_aa 0 0 NA NA NA NA NA NA NA NA NA8.4 Модель «Sturtevant etc» I типу
Ми провели розрахунки, які відповідають робочому циклу моделі, за умови t <- 1. Тепер можна ввімкнути робочий цикл моделі. Для простоти наведемо модель у цілому, причому додамо до неї і останню частину: команду для побудови графіку, який демонструє результати імітації. Таким чином, ось повна модель.
# Sturtevant etc: ВПЛИВ ЧЕРГУВАННЯ ТИПІВ ДОБОРУ НА ХАРАКТЕР ВІДТВОРЕННЯ В ПОПУЛЯЦІЇ
# Модель I типа (однократна імітація)
# I. ENTRANCE — ВХІД:
# I.1. Initial script commands — початкові команди скрипту
# setwd("...") # Робоча директорія
rm(list = ls()) # Очищення раніше збережених об'єктів в Environment
set.seed(12345) # Використання певного варіанту вибору випадкових чисел
# I.2. Initial state of the system — початковий стан модельованої системи
al_0_N_fe <- 100 # Початкова чисельність самиць
al_0_N_ma <- 100 # Початкова чисельність самців
al_0_N_hp <- 0 # Початкова чисельність перехреснозапліднюваних гермафродитів
al_0_N_pg <- 100 # Початкова чисельність партеногенетиків
Genot <- c(0.11, 0.12, 0.12, 0.22) # Початковий розподіл генотипів: 11 = AA, 12 = Aa, 22 = aa
# I.3. Parameters — параметри
K <- 400 # Обмеження ємності середовища
r <- 3 # Плодючість (розмір виводка самиці, гермафродита або партеногенетика)
fe_O_FeMa <- r/2 # Кількість самиць у потомстві самиці (що була запліднена самцем)
ma_O_FeMa <- r/2 # Кількість самців у потомстві самиці (що була запліднена самцем)
hp_O_HpHp <- r # Кількість гермафродитів у потомстві гермафродита (що спарувався з іншим гермафродитом)
pg_O_Pg <- r # Кількість партеногенетиків у потомстві партеногенетика (якому не треба з кимось паруватися)
s <- 0.9 # Сила циклічно змінюваного негативного добору
# I.4. Experimental conditions — умови експерименту з моделлю
cycles <- 28 # Кількість робочих циклів моделі
# II TRANSFORMATIONS SYSTEM CREATION — СТВОРЕННЯ СИСТЕМИ ПЕРЕТВОРЕНЬ:
# II.3. Objects creation — створення об’єктів
rws <- c("Females_AA", "Females_Aa", "Females_aa", "Males_AA", "Males_Aa", "Males_aa", "Hermaph_AA", "Hermaph_Aa", "Hermaph_aa", "Parthen_AA", "Parthen_Aa", "Parthen_aa") # Рядки для матриці з підсумками
cls <- c(0:cycles) # Позначення стовпців для матриці з підсумками
Rez <- matrix(NA, nrow = length(rws), ncol = length(cls), dimnames = list(rws, cls)) # Створення матриці для запису підсумків
Al_Pop <- rep(NA, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg) # Створення вектору для початкової чисельності
# III. CALCULATIONS — РОЗРАХУНКИ:
# III.2. Initial composition creation — утворення початкового складу
if(al_0_N_fe>0) Al_Pop[1:al_0_N_fe] <- 1 # У файл для альфа-чисельності переносяться самиці...
if(al_0_N_ma>0) Al_Pop[(al_0_N_fe+1):(al_0_N_fe+al_0_N_ma)] <- 2 # ... самці...
if(al_0_N_hp>0) Al_Pop[(al_0_N_fe+al_0_N_ma+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp)] <- 3 # ... гермафродити...
if(al_0_N_pg>0) Al_Pop[(al_0_N_fe+al_0_N_ma+al_0_N_hp+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp+al_0_N_pg)] <- 4 # ... партеногенетики
Al_Pop <- Al_Pop + sample(Genot, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg, replace=T) # Випадково визначаються генотипи усіх особин
# III.3. Main work cycle — основний робочий цикл
# III.3.a. Початок основного робочого циклу моделі
for (t in 1:cycles) {
if(t>1) Al_Pop <- na.omit(Om_Pop) # В циклах крім першого Al_Pop - це Om_Pop попереднього циклу без NA
# III.3.b. Чисельність груп на початку циклу підраховується та переноситься у матрицю результатів
Rez["Females_AA", t] <- length(which(Al_Pop==1.11))
Rez["Females_Aa", t] <- length(which(Al_Pop==1.12))
Rez["Females_aa", t] <- length(which(Al_Pop==1.22))
Rez["Males_AA", t] <- length(which(Al_Pop==2.11))
Rez["Males_Aa", t] <- length(which(Al_Pop==2.12))
Rez["Males_aa", t] <- length(which(Al_Pop==2.22))
Rez["Hermaph_AA", t] <- length(which(Al_Pop==3.11))
Rez["Hermaph_Aa", t] <- length(which(Al_Pop==3.12))
Rez["Hermaph_aa", t] <- length(which(Al_Pop==3.22))
Rez["Parthen_AA", t] <- length(which(Al_Pop==4.11))
Rez["Parthen_Aa", t] <- length(which(Al_Pop==4.12))
Rez["Parthen_aa", t] <- length(which(Al_Pop==4.22))
# III.3.d. Розрахунок бета-чисельності (альфа-чисельність + потомство), з ймовірнісним округленням
be_N_Ma <- Rez["Males_AA", t] + Rez["Males_Aa", t] + Rez["Males_aa", t] # Загальна кількість самців
if(be_N_Ma==0) { # Якщо самців нема, бета-чисельність дорівнює альфа-чисельності
be_N_Fe_AA <- Rez["Females_AA", t]
be_N_Fe_Aa <- Rez["Females_Aa", t]
be_N_Fe_aa <- Rez["Females_aa", t]
be_N_Ma_AA <- Rez["Males_AA", t]
be_N_Ma_Aa <- Rez["Males_Aa", t]
be_N_Ma_aa <- Rez["Males_aa", t]
} else { # Якщо самці є, чисельність кожної форми розраховується залежно від схрещувань, де вона може утворюватися
be_N_Fe_AA <- Rez["Females_AA", t] + floor(fe_O_FeMa * # Альфа чисельність + ймовірнісне округлення добутку плодючості та кількості схрещувань, де утворюється потрібний генотип
(Rez["Females_AA", t] * Rez["Males_AA", t] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Fe_Aa <- Rez["Females_Aa", t] + floor(fe_O_FeMa *
(Rez["Females_AA", t] * Rez["Males_aa", t] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t] * Rez["Males_AA", t] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Fe_aa <- Rez["Females_aa", t] + floor(ma_O_FeMa *
(Rez["Females_aa", t] * Rez["Males_aa", t] / be_N_Ma + # aa × aa ⟶ aa
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # aa × Aa ⟶ Aa : aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_AA <- Rez["Males_AA", t] + floor(ma_O_FeMa *
(Rez["Females_AA", t] * Rez["Males_AA", t] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_Aa <- Rez["Males_Aa", t] + floor(ma_O_FeMa *
(Rez["Females_AA", t] * Rez["Males_aa", t] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t] * Rez["Males_AA", t] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_AA", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Ma_aa <- Rez["Males_aa", t] + floor(ma_O_FeMa *
(Rez["Females_aa", t] * Rez["Males_aa", t] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_Aa", t] * Rez["Males_aa", t] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_aa", t] * Rez["Males_Aa", t] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t] * Rez["Males_Aa", t] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
} # Закриття дужок підрахунку самиць та самців після else
be_N_Hp <- Rez["Hermaph_AA", t] + Rez["Hermaph_Aa", t] + Rez["Hermaph_aa", t] # Загальна кількість гермафродитів
if(be_N_Hp<2) { # Якщо гермафродитів менше за два, бета-чисельність дорівнює альфа-чисельності
be_N_Hp_AA <- Rez["Hermaph_AA", t]
be_N_Hp_Aa <- Rez["Hermaph_Aa", t]
be_N_Hp_aa <- Rez["Hermaph_aa", t]
} else { # Розраховується (з окремими позначеннями) чисельність різних за складом пар гермафродитів
if(Rez["Hermaph_AA", t]>1) Pairs_Herms_AA_AA <- Rez["Hermaph_AA", t]*(Rez["Hermaph_AA", t]-1)/be_N_Hp else Pairs_Herms_AA_AA <- 0
if(Rez["Hermaph_Aa", t]>1) Pairs_Herms_Aa_Aa <- Rez["Hermaph_Aa", t]*(Rez["Hermaph_Aa", t]-1)/be_N_Hp else Pairs_Herms_Aa_Aa <- 0
if(Rez["Hermaph_aa", t]>1) Pairs_Herms_aa_aa <- Rez["Hermaph_aa", t]*(Rez["Hermaph_aa", t]-1)/be_N_Hp else Pairs_Herms_aa_aa <- 0
Pairs_Herms_AA_Aa <- Rez["Hermaph_AA", t]*Rez["Hermaph_Aa", t]/be_N_Hp
Pairs_Herms_AA_aa <- Rez["Hermaph_AA", t]*Rez["Hermaph_aa", t]/be_N_Hp
Pairs_Herms_Aa_aa <- Rez["Hermaph_Aa", t]*Rez["Hermaph_aa", t]/be_N_Hp
# А тепер - бета-чисельність різних генотипів залежно від кількості можливих схрещувань
be_N_Hp_AA <- Rez["Hermaph_AA", t] + floor(hp_O_HpHp *
(Pairs_Herms_AA_AA*2 + # AA × AA ⟶ AA
Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_Aa_Aa/2) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Hp_Aa <- Rez["Hermaph_Aa", t] + floor(hp_O_HpHp *
(Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_AA_aa*2 + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_Aa_aa) + runif(1)) # Aa × aa ⟶ Aa : aa
be_N_Hp_aa <- Rez["Hermaph_aa", t] + floor(hp_O_HpHp *
(Pairs_Herms_Aa_aa + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa/2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_aa_aa*2) + runif(1)) # aa × aa ⟶ aa
} # Закриття дужок підрахунку гермафродитів після else
be_N_Pg_AA <- Rez["Parthen_AA", t] + floor(pg_O_Pg * Rez["Parthen_AA", t] + runif(1)) # ...і партеногенетики
be_N_Pg_Aa <- Rez["Parthen_Aa", t] + floor(pg_O_Pg * Rez["Parthen_Aa", t] + runif(1))
be_N_Pg_aa <- Rez["Parthen_aa", t] + floor(pg_O_Pg * Rez["Parthen_aa", t] + runif(1))
# III.3.e. Визначення добору для гомозигот та гетерозигот: залишок від поділу визначає, який йде добір
if(t%%2==1) { # Якщо покоління непарне, то...
v_homozygotes <- 1-s # ...чисельність гомозигот скорочується, ...
v_heterozygotes <- 1 # ...а чисельність гетерозигот лишається постійною
} else { # Якщо покоління парне, то...
v_homozygotes <- 1 # ...чисельність гомозигот лишається постійною...
v_heterozygotes <- 1-s} # ...а чисельність гетерозигот скорочується
# III.3.f. Вплив добору на кожну з груп (розрахунок гама-чисельностей)
ga_N_Fe_AA <- floor(be_N_Fe_AA * v_homozygotes + runif(1)) # Додаток кожної бета-чисельності та...
ga_N_Fe_Aa <- floor(be_N_Fe_Aa * v_heterozygotes + runif(1)) # ...життездатності у даному поколінні...
ga_N_Fe_aa <- floor(be_N_Fe_aa * v_homozygotes + runif(1)) # ...зазнає ймовірнісного округлення:
ga_N_Ma_AA <- floor(be_N_Ma_AA * v_homozygotes + runif(1)) # ...це і є гама-чисельність
ga_N_Ma_Aa <- floor(be_N_Ma_Aa * v_heterozygotes + runif(1))
ga_N_Ma_aa <- floor(be_N_Ma_aa * v_homozygotes + runif(1))
ga_N_Hp_AA <- floor(be_N_Hp_AA * v_homozygotes + runif(1))
ga_N_Hp_Aa <- floor(be_N_Hp_Aa * v_heterozygotes + runif(1))
ga_N_Hp_aa <- floor(be_N_Hp_aa * v_homozygotes + runif(1))
ga_N_Pg_AA <- floor(be_N_Pg_AA * v_homozygotes + runif(1))
ga_N_Pg_Aa <- floor(be_N_Pg_Aa * v_heterozygotes + runif(1))
ga_N_Pg_aa <- floor(be_N_Pg_aa * v_homozygotes + runif(1))
# III.3.g. Створення вектору з гама-складом популяції
Ga_Pop <- c(rep(1.11, ga_N_Fe_AA), rep(1.12, ga_N_Fe_Aa), rep(1.22, ga_N_Fe_aa), rep(2.11, ga_N_Ma_AA), rep(2.12, ga_N_Ma_Aa), rep(2.22, ga_N_Ma_aa), rep(3.11, ga_N_Hp_AA), rep(3.12, ga_N_Hp_Aa), rep(3.22, ga_N_Hp_aa), rep(4.11, ga_N_Pg_AA), rep(4.12, ga_N_Pg_Aa), rep(4.22, ga_N_Pg_aa))
# III.3.h. Створення вектора долі (Fate) і вектора жереба (Lot)
if(length(Ga_Pop)>K) Fate <- rep(NA, length(Ga_Pop)) else Fate <- rep(NA, K) # Визначається довжина вектора долі
Fate[1:K] <- 1 # В векторі долі стільки одиниць, скільки має вижити особин, але вони розташовані впорядковано
Lot <- sample(Fate, length(Ga_Pop), replace = FALSE) # В векторі жереба одиниці та NA випадково переплутані
# III.3.i. Вектор жереба визначає остаточний склад в циклі
Om_Pop <- Ga_Pop * Lot # У векторі Om_Pop лишається K особин; загиблі особини - NA
# III.3.j. Закінчення основного робочого циклу моделі і збереження результатів останнього циклу
} # Основний робочий цикл моделі завершився
Rez["Females_AA", cycles+1] <- length(which(Om_Pop==1.11)) # Заповнення останнього стовпчику у матриці результатів
Rez["Females_Aa", cycles+1] <- length(which(Om_Pop==1.12))
Rez["Females_aa", cycles+1] <- length(which(Om_Pop==1.22))
Rez["Males_AA", cycles+1] <- length(which(Om_Pop==2.11))
Rez["Males_Aa", cycles+1] <- length(which(Om_Pop==2.12))
Rez["Males_aa", cycles+1] <- length(which(Om_Pop==2.22))
Rez["Hermaph_AA", cycles+1] <- length(which(Om_Pop==3.11))
Rez["Hermaph_Aa", cycles+1] <- length(which(Om_Pop==3.12))
Rez["Hermaph_aa", cycles+1] <- length(which(Om_Pop==3.22))
Rez["Parthen_AA", cycles+1] <- length(which(Om_Pop==4.11))
Rez["Parthen_Aa", cycles+1] <- length(which(Om_Pop==4.12))
Rez["Parthen_aa", cycles+1] <- length(which(Om_Pop==4.22))
# IV. FINISHING — ЗАВЕРШЕННЯ:
# IV.3. Results viewing — перегляд підсумків
Rez # Вивід у консоль матриці результатів
## 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17
## Females_AA 18 5 40 7 36 3 26 3 18 1 11 1 7 2 7 1 7 1
## Females_Aa 53 89 17 68 12 54 10 38 7 30 6 16 3 11 2 12 2 3
## Females_aa 29 6 45 4 34 5 29 5 24 2 16 3 12 2 7 0 3 0
## Males_AA 23 3 38 5 35 4 26 2 17 1 11 1 7 1 7 2 8 1
## Males_Aa 47 91 16 70 13 54 10 42 8 24 5 22 4 9 2 9 2 6
## Males_aa 30 7 47 8 38 5 29 5 24 5 20 4 14 1 7 0 3 0
## Hermaph_AA 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## Hermaph_Aa 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## Hermaph_aa 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
## Parthen_AA 22 7 28 8 32 9 36 11 44 14 56 15 60 15 60 18 72 19
## Parthen_Aa 58 185 74 222 89 255 102 286 115 318 127 332 133 353 141 351 140 362
## Parthen_aa 20 7 28 8 32 11 44 8 32 5 20 6 24 6 24 7 28 8
## 18 19 20 21 22 23 24 25 26 27 28
## Females_AA 3 0 1 0 1 0 1 0 1 0 0
## Females_Aa 1 4 1 4 1 4 1 2 0 1 0
## Females_aa 1 1 4 1 4 0 1 0 1 0 0
## Males_AA 3 0 2 0 2 1 2 0 1 0 0
## Males_Aa 1 3 0 4 1 5 1 1 0 0 0
## Males_aa 1 0 3 1 4 1 2 0 1 0 0
## Hermaph_AA 0 0 0 0 0 0 0 0 0 0 0
## Hermaph_Aa 0 0 0 0 0 0 0 0 0 0 0
## Hermaph_aa 0 0 0 0 0 0 0 0 0 0 0
## Parthen_AA 76 19 76 22 88 27 108 22 88 25 100
## Parthen_Aa 145 367 147 361 144 355 142 368 147 369 148
## Parthen_aa 32 6 24 7 28 7 28 7 28 5 20
# IV.4. Results visualization — візуалізація підсумків
plot(Rez["Females_AA", ], type="l", lty=2, col="hotpink", ylim=c(0, K),
main = "«Sturtevant etc»: динаміка в умовах циклічно змінюваного добору\nформ, що відрізняються за способом відтворення та генотипом",
xlab="Цикли імітації", ylab="Чисельність окремих форм")
lines(Rez["Females_Aa", ], type="l", lty=4, col="violetred")
lines(Rez["Females_aa", ], type="l", lty=3, col="magenta")
lines(Rez["Males_AA", ], type="l", lty=2, col="darkslateblue")
lines(Rez["Males_Aa", ], type="l", lty=4, col="royalblue")
lines(Rez["Males_aa", ], type="l", lty=3, col="dodgerblue")
lines(Rez["Hermaph_AA", ], type="l", lty=2, col="seagreen")
lines(Rez["Hermaph_Aa", ], type="l", lty=4, col="darkgreen")
lines(Rez["Hermaph_aa", ], type="l", lty=3, col="olivedrab4")
lines(Rez["Parthen_AA", ], type="l", lty=2, col="sienna")
lines(Rez["Parthen_Aa", ], type="l", lty=4, col="darkorange4")
lines(Rez["Parthen_aa", ], type="l", lty=3, col="tomato4")
legend(12, K, c("Females AA", "Females Aa", "Females aa", "Males AA", "Males Aa", "Males aa", "Hermaphrodites AA", "Hermaphrodites Aa", "Hermaphrodites aa", "Parthenogenetics AA", "Parthenogenetics Aa", "Parthenogenetics aa"), col = c("hotpink", "violetred", "magenta", "darkslateblue", "royalblue", "dodgerblue", "seagreen", "darkgreen", "olivedrab4", "sienna", "darkorange4", "tomato4"), lty = c(2, 4, 3, 2, 4, 3, 2, 4, 3, 2, 4, 3))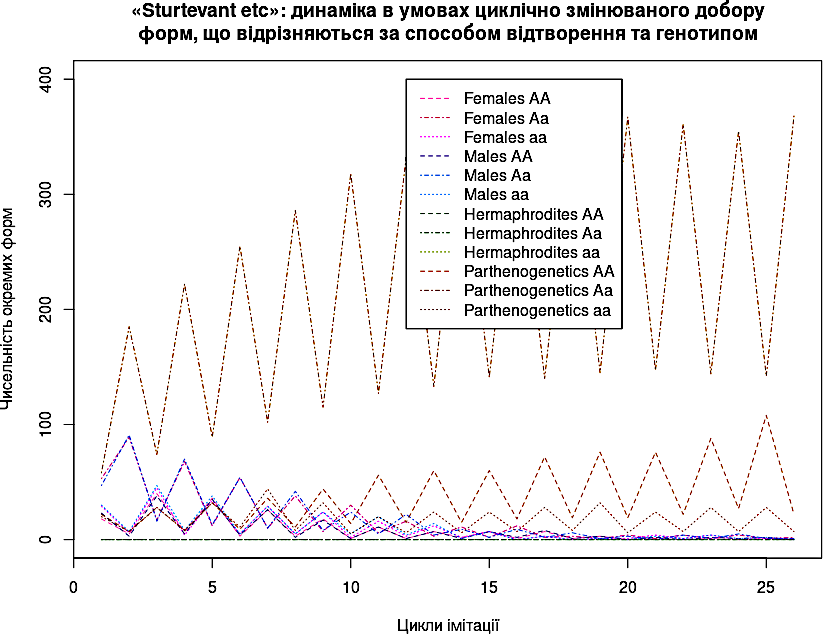
Рис. 8.4.1 Результат роботи наведеної вище моделі
На графіку колір лінії пов’язаний з типом відтворення (самиці — червоні, самці — блакитні, гермафродити — зелені, партеногенетики — коричневі), а генотип — з характером лінії, який задається параметром lty (AA — пунктир з відносно більших, а aa — з відносно менших відтінів, а Aa — пунктир з чергуванням більших та менших відтінків).
Модель працює! Лишилося найголовніше: зрозуміти, які висновки можна зробити з її допомогою. Ймовірно, для цього буде необхідним побудувати на базі цієї моделі I типу наступні — II та III типів.
Зверніть увагу: у показаному на рис. 8.4.1 випадку діє дуже суттєвий циклічно змінюваний добір: s=0.9! У кожному поколінні знищується 90% або гомозигот, або гетерозигот. І, усе ж таки, до 28-го покоління партеногенетики повністю витісняють своїх роздільностатевих конкурентів! Ситуація далеко не така проста, як може здаватися...
8.5 Модель «Sturtevant etc» II типу
Після того, як ми зробили модель, яка коректно (здається) проводить одну імітацію, слід навчитися проводити низку імітацій.
Перший розділ скрипту (I. ENTRANCE — ВХІД) може лишитися без змін, за виключенням одного рядку, який зазначає кількість імітацій.
Результати роботи моделі відбивалися у матриці Rez; її рядки — форми особин, що відрізняються за розмноженням та генотипами, а стовпці — цикли. Якщо ми додаємо групу імітацій, нам потрібний ще один вимір. Тому замість двовимірної матриці створимо тривимірний масив (array).
Як аналізувати результати групи ітерацій? Створимо три вектори (FM, H, P) де будемо збирати дані про остаточний склад модельної популяції, а також вектор Types, у якому будемо визначати, які типи за характером відтворення лишилися в цій популяції.
Розділ III (CALCULATIONS — РОЗРАХУНКИ) скрипту слід починати з циклу ітерацій. Далі основну частину усього цього великого розділу можна залишити без змін, і лише наприкінці додати фрагмент, який буде створювати вектори для аналізу результатів.
Як позначати кількість особин певної форми? Як їх частку від загальної кількості. Це викликає небезпеку поділу на 0 у разі, якщо модельна популяція загине. Тому доведеться використати функцію if() {} else {}. Якщо усі особини зникли, у вектори FM, H та P слід без усяких додаткових розрахунків вписати нулі. Якщо хтось лишився, слід розрахувати частки особин з різним типом розмноження, а потім застосувати низку умов, які дозволять автоматично визначити тип модельної популяції (передбачені варіанти — FM, H, P, FM_H, FM_P, H_P, FM_H_P). Після цього лишилося тільки закрити цикл ітерацій.
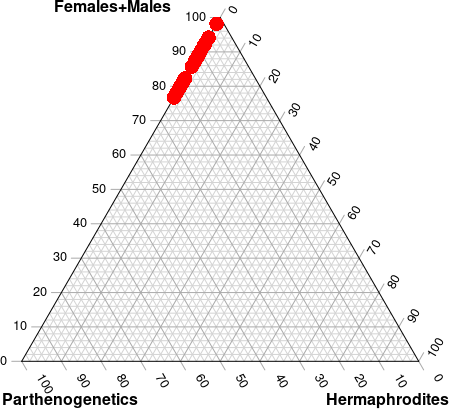
Щоб оцінити результати сукупності ітерацій, можна просто вивести в консоль вектор Types, а можна шукати адекватні способи візуалізації. Спробуємо використати достатньо незвичний тип візуалізації результатів: тернарну діаграму. Це — досить красивий двовимірний засіб візуалізації розташування точок у просторі трьох координат. Координати кожної точки визначають її проекції на усі три осі; ці проекції слід робити за прямими, які паралельні осям. Для побудови тернарного графіку використаємо бібліотеку Ternary. Звісно, при першому застосуванні слід інсталювати цей пакет. Входом для нього є об’єкти формату data.frame; такий об’єкт (під назвою Resume).
Можна впевнитися, що у разі наявності в моделі гермафродитів, вони закономірно перемагають інші форми відтворення. Якщо гермафродитів в модельній популяції нема, результати моделювання стають менш прогнозованими. З цієї точки зору тернарні графіки — не найкраще рішення саме для цього випадку, адже тривимірна картинка швидко перетворюється на двовимірну.
Наведемо текст моделі II типу повністю.
# Sturtevant etc: ВПЛИВ ЧЕРГУВАННЯ ТИПІВ ДОБОРУ НА ХАРАКТЕР ВІДТВОРЕННЯ В ПОПУЛЯЦІЇ
# Модель II типа (аналіз розподілу імовірностей результатів для певних початкових умов)
# I. ENTRANCE — ВХІД:
# I.1. Initial script commands — початкові команди скрипту
setwd("~/!_Courses/Sex_on_R") # Робоча директорія (лише на комп'ютері Д.Ш.!!!)
rm(list = ls()) # Очищення раніше збережених об'єктів в Environment
# set.seed(12345) # Використання певного варіанту вибору випадкових чисел
# I.2. Initial state of the system — початковий стан модельованої системи
al_0_N_fe <- 150 # Початкова чисельність самиць
al_0_N_ma <- 150 # Початкова чисельність самців
al_0_N_hp <- 150 # Початкова чисельність перехреснозапліднюваних гермафродитів
al_0_N_pg <- 150 # Початкова чисельність партеногенетиків
Genot <- c(0.11, 0.12, 0.12, 0.22) # Початковий розподіл генотипів: 11 = AA, 12 = Aa, 22 = aa
# I.3. Parameters — параметри
K <- 600 # Обмеження ємності середовища
r <- 8 # Плодючість (розмір виводка самиці, гермафродита або партеногенетика)
fe_O_FeMa <- r/2 # Кількість самиць у потомстві самиці (що була запліднена самцем)
ma_O_FeMa <- r/2 # Кількість самців у потомстві самиці (що була запліднена самцем)
hp_O_HpHp <- r # Кількість гермафродитів у потомстві гермафродита (що спарувався з іншим гермафродитом)
pg_O_Pg <- r # Кількість партеногенетиків у потомстві партеногенетика (якому не треба з кимось паруватися)
s <- 0.94 # Сила циклічно змінюваного негативного добору
# I.4. Experimental conditions — умови експерименту з моделлю
cycles <- 50 # Кількість робочих циклів моделі
iterat <- 20 # Кількість ітерацій
# II TRANSFORMATIONS SYSTEM CREATION — СТВОРЕННЯ СИСТЕМИ ПЕРЕТВОРЕНЬ:
# II.3. Objects creation — створення об’єктів
rws <- c("Females_AA", "Females_Aa", "Females_aa", "Males_AA", "Males_Aa", "Males_aa", "Hermaph_AA", "Hermaph_Aa", "Hermaph_aa", "Parthen_AA", "Parthen_Aa", "Parthen_aa") # Рядки для масиву з підсумками
cls <- c(0:cycles) # Позначення стовпців для масиву з підсумками
itr <- 1:iterat # Створення вектору, куди будуть записані "імена" ітерацій
for (d in 1:iterat){First <- "Iteration_"; Second <- d; itr[d] <- paste0(First, Second) } # Iterations names
Rez <- array(NA, dim = c(length(rws), length(cls), length(itr)), dimnames = list(rws, cls, itr)) # Створення тривимірного масиву для запису підсумків
FM <- rep(NA, iterat); H <- rep(NA, iterat); P <- rep(NA, iterat); Types <- rep(NA, iterat)
Al_Pop <- rep(NA, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg) # Створення вектору для початкової чисельності
# III. CALCULATIONS — РОЗРАХУНКИ:
# III.1. Higher level cycles running — запуск циклів вищого рівня
for (i in 1:iterat) { # Початок ітерації
# III.2. Initial composition creation — утворення початкового складу
if(al_0_N_fe>0) Al_Pop[1:al_0_N_fe] <- 1 # У файл для альфа-чисельності переносяться самиці...
if(al_0_N_ma>0) Al_Pop[(al_0_N_fe+1):(al_0_N_fe+al_0_N_ma)] <- 2 # ... самці...
if(al_0_N_hp>0) Al_Pop[(al_0_N_fe+al_0_N_ma+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp)] <- 3 # ... гермафродити...
if(al_0_N_pg>0) Al_Pop[(al_0_N_fe+al_0_N_ma+al_0_N_hp+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp+al_0_N_pg)] <- 4 # ... партеногенетики
Al_Pop <- Al_Pop + sample(Genot, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg, replace=T) # Випадково визначаються генотипи усіх особин
# III.3. Main work cycle — основний робочий цикл
# III.3.a. Початок основного робочого циклу моделі
for (t in 1:cycles) {
if(t>1) Al_Pop <- na.omit(Om_Pop) # В циклах крім першого Al_Pop - це Om_Pop попереднього циклу без NA
# III.3.b. Чисельність груп на початку циклу підраховується та переноситься у масив з результатами
Rez["Females_AA", t, i] <- length(which(Al_Pop==1.11))
Rez["Females_Aa", t, i] <- length(which(Al_Pop==1.12))
Rez["Females_aa", t, i] <- length(which(Al_Pop==1.22))
Rez["Males_AA", t, i] <- length(which(Al_Pop==2.11))
Rez["Males_Aa", t, i] <- length(which(Al_Pop==2.12))
Rez["Males_aa", t, i] <- length(which(Al_Pop==2.22))
Rez["Hermaph_AA", t, i] <- length(which(Al_Pop==3.11))
Rez["Hermaph_Aa", t, i] <- length(which(Al_Pop==3.12))
Rez["Hermaph_aa", t, i] <- length(which(Al_Pop==3.22))
Rez["Parthen_AA", t, i] <- length(which(Al_Pop==4.11))
Rez["Parthen_Aa", t, i] <- length(which(Al_Pop==4.12))
Rez["Parthen_aa", t, i] <- length(which(Al_Pop==4.22))
# III.3.d. Розрахунок бета-чисельності (альфа-чисельність + потомство), з ймовірнісним округленням
be_N_Ma <- Rez["Males_AA", t, i] + Rez["Males_Aa", t, i] + Rez["Males_aa", t, i] # Загальна кількість самців
if(be_N_Ma==0) { # Якщо самців нема, бета-чисельність дорівнює альфа-чисельності
be_N_Fe_AA <- Rez["Females_AA", t, i]
be_N_Fe_Aa <- Rez["Females_Aa", t, i]
be_N_Fe_aa <- Rez["Females_aa", t, i]
be_N_Ma_AA <- Rez["Males_AA", t, i]
be_N_Ma_Aa <- Rez["Males_Aa", t, i]
be_N_Ma_aa <- Rez["Males_aa", t, i]
} else { # Якщо самці є, чисельність кожної форми розраховується залежно від схрещувань, де вона може утворюватися
be_N_Fe_AA <- Rez["Females_AA", t, i] + floor(fe_O_FeMa * # Альфа чисельність + ймовірнісне округлення добутку плодючості та кількості схрещувань, де утворюється потрібний генотип
(Rez["Females_AA", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Fe_Aa <- Rez["Females_Aa", t, i] + floor(fe_O_FeMa *
(Rez["Females_AA", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Fe_aa <- Rez["Females_aa", t, i] + floor(ma_O_FeMa *
(Rez["Females_aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # aa × aa ⟶ aa
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # aa × Aa ⟶ Aa : aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_AA <- Rez["Males_AA", t, i] + floor(ma_O_FeMa *
(Rez["Females_AA", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_Aa <- Rez["Males_Aa", t, i] + floor(ma_O_FeMa *
(Rez["Females_AA", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Ma_aa <- Rez["Males_aa", t, i] + floor(ma_O_FeMa *
(Rez["Females_aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
} # Закриття дужок підрахунку самиць та самців після else
be_N_Hp <- Rez["Hermaph_AA", t, i] + Rez["Hermaph_Aa", t, i] + Rez["Hermaph_aa", t, i] # Загальна кількість гермафродитів
if(be_N_Hp<2) { # Якщо гермафродитів менше за два, бета-чисельність дорівнює альфа-чисельності
be_N_Hp_AA <- Rez["Hermaph_AA", t, i]
be_N_Hp_Aa <- Rez["Hermaph_Aa", t, i]
be_N_Hp_aa <- Rez["Hermaph_aa", t, i]
} else { # Розраховується (з окремими позначеннями) чисельність різних за складом пар гермафродитів
if(Rez["Hermaph_AA", t, i]>1) Pairs_Herms_AA_AA <- Rez["Hermaph_AA", t, i]*(Rez["Hermaph_AA", t, i]-1)/be_N_Hp else Pairs_Herms_AA_AA <- 0
if(Rez["Hermaph_Aa", t, i]>1) Pairs_Herms_Aa_Aa <- Rez["Hermaph_Aa", t, i]*(Rez["Hermaph_Aa", t, i]-1)/be_N_Hp else Pairs_Herms_Aa_Aa <- 0
if(Rez["Hermaph_aa", t, i]>1) Pairs_Herms_aa_aa <- Rez["Hermaph_aa", t, i]*(Rez["Hermaph_aa", t, i]-1)/be_N_Hp else Pairs_Herms_aa_aa <- 0
Pairs_Herms_AA_Aa <- Rez["Hermaph_AA", t, i]*Rez["Hermaph_Aa", t, i]/be_N_Hp
Pairs_Herms_AA_aa <- Rez["Hermaph_AA", t, i]*Rez["Hermaph_aa", t, i]/be_N_Hp
Pairs_Herms_Aa_aa <- Rez["Hermaph_Aa", t, i]*Rez["Hermaph_aa", t, i]/be_N_Hp
# А тепер - бета-чисельність різних генотипів залежно від кількості можливих схрещувань
be_N_Hp_AA <- Rez["Hermaph_AA", t, i] + floor(hp_O_HpHp *
(Pairs_Herms_AA_AA*2 + # AA × AA ⟶ AA
Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_Aa_Aa/2) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Hp_Aa <- Rez["Hermaph_Aa", t, i] + floor(hp_O_HpHp *
(Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_AA_aa*2 + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_Aa_aa) + runif(1)) # Aa × aa ⟶ Aa : aa
be_N_Hp_aa <- Rez["Hermaph_aa", t, i] + floor(hp_O_HpHp *
(Pairs_Herms_Aa_aa + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa/2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_aa_aa*2) + runif(1)) # aa × aa ⟶ aa
} # Закриття дужок підрахунку гермафродитів після else
be_N_Pg_AA <- Rez["Parthen_AA", t, i] + floor(pg_O_Pg * Rez["Parthen_AA", t, i] + runif(1)) # ...і партеногенетики
be_N_Pg_Aa <- Rez["Parthen_Aa", t, i] + floor(pg_O_Pg * Rez["Parthen_Aa", t, i] + runif(1))
be_N_Pg_aa <- Rez["Parthen_aa", t, i] + floor(pg_O_Pg * Rez["Parthen_aa", t, i] + runif(1))
# III.3.e. Визначення добору для гомозигот та гетерозигот: залишок від поділу визначає, який йде добір
if(t%%2==1) { # Якщо покоління непарне, то...
v_homozygotes <- 1-s # ...чисельність гомозигот скорочується, ...
v_heterozygotes <- 1 # ...а чисельність гетерозигот лишається постійною
} else { # Якщо покоління парне, то...
v_homozygotes <- 1 # ...чисельність гомозигот лишається постійною...
v_heterozygotes <- 1-s} # ...а чисельність гетерозигот скорочується
# III.3.f. Вплив добору на кожну з груп (розрахунок гама-чисельностей)
ga_N_Fe_AA <- floor(be_N_Fe_AA * v_homozygotes + runif(1)) # Додаток кожної бета-чисельності та...
ga_N_Fe_Aa <- floor(be_N_Fe_Aa * v_heterozygotes + runif(1)) # ...життездатності у даному поколінні...
ga_N_Fe_aa <- floor(be_N_Fe_aa * v_homozygotes + runif(1)) # ...зазнає ймовірнісного округлення:
ga_N_Ma_AA <- floor(be_N_Ma_AA * v_homozygotes + runif(1)) # ...це і є гама-чисельність
ga_N_Ma_Aa <- floor(be_N_Ma_Aa * v_heterozygotes + runif(1))
ga_N_Ma_aa <- floor(be_N_Ma_aa * v_homozygotes + runif(1))
ga_N_Hp_AA <- floor(be_N_Hp_AA * v_homozygotes + runif(1))
ga_N_Hp_Aa <- floor(be_N_Hp_Aa * v_heterozygotes + runif(1))
ga_N_Hp_aa <- floor(be_N_Hp_aa * v_homozygotes + runif(1))
ga_N_Pg_AA <- floor(be_N_Pg_AA * v_homozygotes + runif(1))
ga_N_Pg_Aa <- floor(be_N_Pg_Aa * v_heterozygotes + runif(1))
ga_N_Pg_aa <- floor(be_N_Pg_aa * v_homozygotes + runif(1))
# III.3.g. Створення вектору з гама-складом популяції
Ga_Pop <- c(rep(1.11, ga_N_Fe_AA), rep(1.12, ga_N_Fe_Aa), rep(1.22, ga_N_Fe_aa), rep(2.11, ga_N_Ma_AA), rep(2.12, ga_N_Ma_Aa), rep(2.22, ga_N_Ma_aa), rep(3.11, ga_N_Hp_AA), rep(3.12, ga_N_Hp_Aa), rep(3.22, ga_N_Hp_aa), rep(4.11, ga_N_Pg_AA), rep(4.12, ga_N_Pg_Aa), rep(4.22, ga_N_Pg_aa))
# III.3.h. Створення вектора долі (Fate) і вектора жереба (Lot)
if(length(Ga_Pop)>K) Fate <- rep(NA, length(Ga_Pop)) else Fate <- rep(NA, K) # Визначається довжина вектора долі
Fate[1:K] <- 1 # В векторі долі стільки одиниць, скільки має вижити особин, але вони розташовані впорядковано
Lot <- sample(Fate, length(Ga_Pop), replace = FALSE) # В векторі жереба одиниці та NA випадково переплутані
# III.3.i. Вектор жереба визначає остаточний склад в циклі
Om_Pop <- Ga_Pop * Lot # У векторі Om_Pop лишається K особин; загиблі особини - NA
# III.3.j. Закінчення основного робочого циклу моделі і збереження результатів останнього циклу
} # Основний робочий цикл моделі завершився
Rez["Females_AA", cycles+1, i] <- length(which(Om_Pop==1.11)) # Заповнення останнього стовпчику для результатів даної ітерації
Rez["Females_Aa", cycles+1, i] <- length(which(Om_Pop==1.12))
Rez["Females_aa", cycles+1, i] <- length(which(Om_Pop==1.22))
Rez["Males_AA", cycles+1, i] <- length(which(Om_Pop==2.11))
Rez["Males_Aa", cycles+1, i] <- length(which(Om_Pop==2.12))
Rez["Males_aa", cycles+1, i] <- length(which(Om_Pop==2.22))
Rez["Hermaph_AA", cycles+1, i] <- length(which(Om_Pop==3.11))
Rez["Hermaph_Aa", cycles+1, i] <- length(which(Om_Pop==3.12))
Rez["Hermaph_aa", cycles+1, i] <- length(which(Om_Pop==3.22))
Rez["Parthen_AA", cycles+1, i] <- length(which(Om_Pop==4.11))
Rez["Parthen_Aa", cycles+1, i] <- length(which(Om_Pop==4.12))
Rez["Parthen_aa", cycles+1, i] <- length(which(Om_Pop==4.22))
if(sum(Rez[, t+1, i])==0) {FM[i] <- 0; H[i] <- 0; P[i] <- 0} else { # заповнення вектора Resume
FM[i] <- sum(Rez[1:6, t+1, i])/sum(Rez[, t+1, i])
H[i] <- sum(Rez[7:9, t+1, i])/sum(Rez[, t+1, i])
P[i] <- sum(Rez[10:12, t+1, i])/sum(Rez[, t+1, i])
if (H[i]==0 & P[i]==0) Types[i] <- "FM"
if (FM[i]==0 & P[i]==0) Types[i] <- "H"
if (FM[i]==0 & H[i]==0) Types[i] <- "P"
if (FM[i]>0 & H[i]>0 & P[i]==0) Types[i] <- "FM_H"
if (FM[i]>0 & H[i]==0 & P[i]>0) Types[i] <- "FM_P"
if (FM[i]==0 & H[i]>0 & P[i]>0) Types[i] <- "H_P"
if (FM[i]>0 & H[i]>0 & P[i]>0) Types[i] <- "FM_H_P"
} # кінець else
# III.4. Ending of higher level cycles — завершення циклів вищого рівня
} # Кінець циклу ітерацій
# IV. FINISHING — ЗАВЕРШЕННЯ:
# IV.3. Results viewing — перегляд підсумків
Resume <- data.frame(FM, H, P)
Resume
## FM H P
## 1 0 1 0
## 2 0 1 0
## 3 0 1 0
## 4 0 1 0
## 5 0 1 0
## 6 0 1 0
## 7 0 1 0
## 8 0 1 0
## 9 0 1 0
## 10 0 1 0
## 11 0 1 0
## 12 0 1 0
## 13 0 1 0
## 14 0 1 0
## 15 0 1 0
## 16 0 1 0
## 17 0 1 0
## 18 0 1 0
## 19 0 1 0
## 20 0 1 0
# Вивід у консоль масиву результатів
Types
## [1] "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H" "H"
## [20] "H"
# IV.4. Results visualization — візуалізація підсумків
# install.packages("Ternary")
library(Ternary)
## This version of 'bslib' is designed to work with 'shiny' >= 1.6.0.
## Please upgrade via install.packages('shiny').
TernaryPlot("Females+Males", "Hermaphrodites", "Parthenogenetics")
TernaryPoints(Resume, pch = 16, cex = 2, col = "red")
В модельній популяції були гермафродити; лишилися тільки вони. А якщо гермафродитів не буде? Використаємо наступні початкові чисельності, і залишимо усе інше без змін.
al_0_N_fe <- 200 # Початкова чисельність самиць
al_0_N_ma <- 200 # Початкова чисельність самців
al_0_N_hp <- 0 # Початкова чисельність перехреснозапліднюваних гермафродитів
al_0_N_pg <- 200 # Початкова чисельність партеногенетиківРезультат є більш складним.
Настав час будувати ще складнішу модель…
8.6 Модель «Sturtevant etc» III типу
Які зміни необхідні для перебудови моделі у III тип? Включимо в початкові команди виклик пакету plot3D. Не будемо задавати параметри r та s, адже вони будуть змінюватися. Наслідком цього стає те, що рядки, де визначатиметься плодючість різних форм, слід перемістити кудись всередину циклу, що змінює r.
Тепер є необхідним новий пункт (I.5. The changeable parameters combinations — комбінації змінюваних параметрів), де будуть задані ті значення названих параметрів, які ми будемо перебирати. Створимо тривимірний масив Outcome. Три «шари» цього масиву відповідатимуть трьом формам відтворення; один з цих «шарів» можна буде використовувати для побудови тривимірної діаграми за допомогою пакету plot3D. Побудова векторів вектори FM, H, P та Types втрачає сенс; ці команди можна прибрати.
Тепер можна почати цикли, що змінюють r та s. Після початку циклу зміни r можна додати команду, яка відбиватиме у консолі прогрес розрахунків, а також визначення плодючості різних форм.
Далі можна привести основну частину розрахунків з мінімальними змінами. Масив Rez буде створюватися у кожному «пакеті» ітерацій (хоча можна було і перенести його вище у скрипті і створювати ще до того, як будуть запущені цикли перебору параметрів).
Як можна побачити, ця модель може вимагати достатньо великої кількості розрахунків. При деяких сполученнях параметрів тривалі розрахунки не мають великого сенсу. тому є сенс вставити у скримп наступні рядки.
# III.3.j. Закінчення основного робочого циклу моделі і збереження результатів останнього циклу
FM <- length(which(Om_Pop<3)) # Перевірка кількості трьох форм відтворення...
Hp <- length(which(Om_Pop<4))-FM #...щоб у разі, коли залишається тільки одна...
Pg <- length(which(Om_Pop<5))-Hp # ...перервати цикл та зекономити час
if(FM==0 & Hp==0 | FM==0 & Pg==0 | Hp==0 & Pg==0) break # умова переривання даної ітераціїЯк це працює? Різницею за генотипами представників різних форм можна знехтувати. Найпростіший спосіб підрахувати кількість самиць та самців (разом узятих) — підрахувати кількість кодів окремих особин, які менші за 3. Ті коди, що менші за 4, але не відповідають ані самицям, ані самцям, відповідають гермафродитам.
Далі треба просто зробити перевірку, чи скінчилися в модельній популяції представники хоча б двох з трьох можливих форм відтворення. В ній використані символи & (І, поєднують умови, що мають бути виконані водночас) та | (АБО, поєднує умови, з яких має бути виконана хоча б одна). У разі виконання цієї умови команда break зупинить цикл, в якому відтворюється. Найнижчим циклом, що виконується на цей момент виконання скрипту, є основний робочий цикл моделі. Це зупинить цикли всередині певної ітерації.
Щоб результати перерваної ітерації були враховані, після її переривання відбувається збереження отриманих кількостей у стовпчики cycles+1 масиву Rez.
Після закінчення пакету імітацій відбувається заповнення відповідних комірок масиву Outcome. Розрахунок робиться так, щоб для кожної з форм вказувати її частку від загальної чисельності.
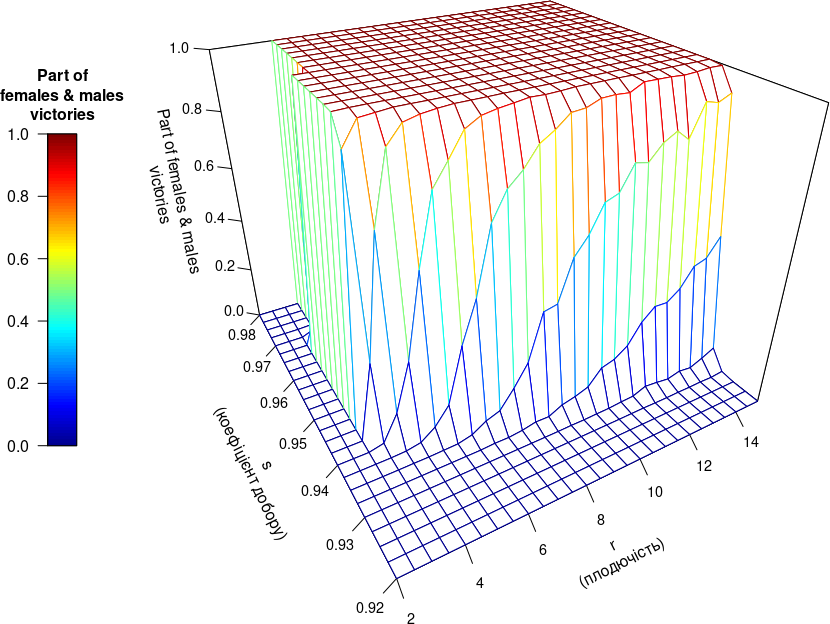
Outcome[a, b, "Females+Males"] <- sum(Rez[1:6, cycles+1, ])/(K*iterat)
Outcome[a, b, "Hermaphrodites"] <- sum(Rez[7:9, cycles+1, ])/(K*iterat)
Outcome[a, b, "Parthenogenetics"] <- sum(Rez[10:12, cycles+1, ])/(K*iterat)Далі залишиться побудувати діаграму, яка буде інтегрувати роботу моделі III типу в цілому.
Тепер можна навести модель III типу в цілому.
# Sturtevant etc: ВПЛИВ ЧЕРГУВАННЯ ТИПІВ ДОБОРУ НА ХАРАКТЕР ВІДТВОРЕННЯ В ПОПУЛЯЦІЇ
# Модель III типа (аналіз впливу змінюваних початкових параметрів на розподіл імовірностей результатів)
# I. ENTRANCE — ВХІД:
# I.1. Initial script commands — початкові команди скрипту
# install.packages("plot3D")
library(plot3D)
# setwd("~/!_Courses/Sex_on_R") # Робоча директорія (лише на комп'ютері Д.Ш.!!!)
rm(list = ls()) # Очищення раніше збережених об'єктів в Environment
# set.seed(12345) # Використання певного варіанту вибору випадкових чисел
# I.2. Initial state of the system — початковий стан модельованої системи
al_0_N_fe <- 200 # Початкова чисельність самиць
al_0_N_ma <- 200 # Початкова чисельність самців
al_0_N_hp <- 0 # Початкова чисельність перехреснозапліднюваних гермафродитів
al_0_N_pg <- 200 # Початкова чисельність партеногенетиків
Genot <- c(0.11, 0.12, 0.12, 0.22) # Початковий розподіл генотипів: 11 = AA, 12 = Aa, 22 = aa
# I.3. Parameters — параметри
K <- 600 # Обмеження ємності середовища
# r <- 4 # ЗМІНЮВАНИЙ ПАРАМЕТР 1: Параметр Мальтуса, плодючість (розмір виводка)
# s <- 0.99 # ЗМІНЮВАНИЙ ПАРАМЕТР 2: Сила циклічно змінюваного негативного добору
# I.4. Experimental conditions — умови експерименту з моделлю
cycles <- 200 # Кількість робочих циклів моделі
iterat <- 100 # Кількість ітерацій
# I.5. The changeable parameters combinations — комбінації змінюваних параметрів
vector_r <- c(2, 2.5, 3, 3.5, 4, 4.5, 5, 5.5, 6, 6.5, 7, 7.5, 8, 8.5, 9, 9.5, 10, 10.5, 11, 11.5, 12, 12.5, 13, 13.5, 14, 14.5, 15)
vector_s <- c(0.92, 0.9225, 0.925, 0.9275, 0.93, 0.9325, 0.935, 0.9375, 0.94, 0.9425, 0.945, 0.9475, 0.95, 0.9525, 0.955, 0.9575, 0.96, 0.9625, 0.965, 0.9675, 0.97, 0.9725, 0.975, 0.9775, 0.98)
# II TRANSFORMATIONS SYSTEM CREATION — СТВОРЕННЯ СИСТЕМИ ПЕРЕТВОРЕНЬ:
# II.2. Parameters combinations mechanism — механізм комбінації змінюваних параметрів
ReproForms <- c("Females+Males", "Hermaphrodites", "Parthenogenetics")
Outcome <- array(NA, dim = c(length(vector_r), length(vector_s), length(ReproForms)), dimnames = list(vector_r, vector_s, ReproForms))
# Цикли, що перебирають досліджувані сполучення параметрів:
for (a in 1:length(vector_r)){ # Цикл, що змінює значення параметру Мальтуса
r <- vector_r[a] # Змінюване значення чисельності популяції
fe_O_FeMa <- r/2 # Кількість самиць у потомстві самиці (що була запліднена самцем)
ma_O_FeMa <- r/2 # Кількість самців у потомстві самиці (що була запліднена самцем)
hp_O_HpHp <- r # Кількість гермафродитів у потомстві гермафродита (що спарувався з іншим гермафродитом)
pg_O_Pg <- r # Кількість партеногенетиків у потомстві партеногенетика (якому не треба з кимось паруватися)
print(paste(a, "from", length(vector_r))) # Виводить у консоль кількість виконаних циклів вищого рівня
for (b in 1:length(vector_s)){ # Цикл, що змінює максимальну тривалість життя
s <- vector_s[b] # Змінюване значення сили добору
# II.3. Objects creation — створення об’єктів
rws <- c("Females_AA", "Females_Aa", "Females_aa", "Males_AA", "Males_Aa", "Males_aa", "Hermaph_AA", "Hermaph_Aa", "Hermaph_aa", "Parthen_AA", "Parthen_Aa", "Parthen_aa") # Рядки для масиву з підсумками
cls <- c(0:cycles) # Позначення стовпців для масиву з підсумками
itr <- 1:iterat # Створення вектору, куди будуть записані "імена" ітерацій
for (d in 1:iterat){First <- "Iteration_"; Second <- d; itr[d] <- paste0(First, Second) } # Iterations names
Rez <- array(NA, dim = c(length(rws), length(cls), length(itr)), dimnames = list(rws, cls, itr)) # Створення тривимірного масиву для запису підсумків
Al_Pop <- rep(NA, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg) # Створення вектору для початкової чисельності
# III. CALCULATIONS — РОЗРАХУНКИ:
# III.1. Higher level cycles running — запуск циклів вищого рівня
for (i in 1:iterat) { # Початок ітерації
# III.2. Initial composition creation — утворення початкового складу
if(al_0_N_fe>0) Al_Pop[1:al_0_N_fe] <- 1 # У файл для альфа-чисельності переносяться самиці...
if(al_0_N_ma>0) Al_Pop[(al_0_N_fe+1):(al_0_N_fe+al_0_N_ma)] <- 2 # ... самці...
if(al_0_N_hp>0) Al_Pop[(al_0_N_fe+al_0_N_ma+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp)] <- 3 # ... гермафродити...
if(al_0_N_pg>0) Al_Pop[(al_0_N_fe+al_0_N_ma+al_0_N_hp+1):(al_0_N_fe+al_0_N_ma+al_0_N_hp+al_0_N_pg)] <- 4 # ... партеногенетики
Al_Pop <- Al_Pop + sample(Genot, al_0_N_fe + al_0_N_ma + al_0_N_hp + al_0_N_pg, replace=T) # Випадково визначаються генотипи усіх особин
# III.3. Main work cycle — основний робочий цикл
# III.3.a. Початок основного робочого циклу моделі
for (t in 1:cycles) {
if(t>1) Al_Pop <- na.omit(Om_Pop) # В циклах крім першого Al_Pop - це Om_Pop попереднього циклу без NA
# III.3.b. Чисельність груп на початку циклу підраховується та переноситься у масив з результатами
Rez["Females_AA", t, i] <- length(which(Al_Pop==1.11))
Rez["Females_Aa", t, i] <- length(which(Al_Pop==1.12))
Rez["Females_aa", t, i] <- length(which(Al_Pop==1.22))
Rez["Males_AA", t, i] <- length(which(Al_Pop==2.11))
Rez["Males_Aa", t, i] <- length(which(Al_Pop==2.12))
Rez["Males_aa", t, i] <- length(which(Al_Pop==2.22))
Rez["Hermaph_AA", t, i] <- length(which(Al_Pop==3.11))
Rez["Hermaph_Aa", t, i] <- length(which(Al_Pop==3.12))
Rez["Hermaph_aa", t, i] <- length(which(Al_Pop==3.22))
Rez["Parthen_AA", t, i] <- length(which(Al_Pop==4.11))
Rez["Parthen_Aa", t, i] <- length(which(Al_Pop==4.12))
Rez["Parthen_aa", t, i] <- length(which(Al_Pop==4.22))
# III.3.d. Розрахунок бета-чисельності (альфа-чисельність + потомство), з ймовірнісним округленням
be_N_Ma <- Rez["Males_AA", t, i] + Rez["Males_Aa", t, i] + Rez["Males_aa", t, i] # Загальна кількість самців
if(be_N_Ma==0) { # Якщо самців нема, бета-чисельність дорівнює альфа-чисельності
be_N_Fe_AA <- Rez["Females_AA", t, i]
be_N_Fe_Aa <- Rez["Females_Aa", t, i]
be_N_Fe_aa <- Rez["Females_aa", t, i]
be_N_Ma_AA <- Rez["Males_AA", t, i]
be_N_Ma_Aa <- Rez["Males_Aa", t, i]
be_N_Ma_aa <- Rez["Males_aa", t, i]
} else { # Якщо самці є, чисельність кожної форми розраховується залежно від схрещувань, де вона може утворюватися
be_N_Fe_AA <- Rez["Females_AA", t, i] + floor(fe_O_FeMa * # Альфа чисельність + ймовірнісне округлення добутку плодючості та кількості схрещувань, де утворюється потрібний генотип
(Rez["Females_AA", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Fe_Aa <- Rez["Females_Aa", t, i] + floor(fe_O_FeMa *
(Rez["Females_AA", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Fe_aa <- Rez["Females_aa", t, i] + floor(ma_O_FeMa *
(Rez["Females_aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # aa × aa ⟶ aa
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # aa × Aa ⟶ Aa : aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_AA <- Rez["Males_AA", t, i] + floor(ma_O_FeMa *
(Rez["Females_AA", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Ma_Aa <- Rez["Males_Aa", t, i] + floor(ma_O_FeMa *
(Rez["Females_AA", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # AA × aa ⟶ Aa
Rez["Females_aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma + # aa × AA ⟶ Aa
Rez["Females_AA", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_AA", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # Aa × aa ⟶ Aa : aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2) + runif(1)) # aa × Aa ⟶ Aa : aa
be_N_Ma_aa <- Rez["Males_aa", t, i] + floor(ma_O_FeMa *
(Rez["Females_aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma + # AA × AA ⟶ AA
Rez["Females_Aa", t, i] * Rez["Males_aa", t, i] / be_N_Ma / 2 + # AA × Aa ⟶ AA : Aa
Rez["Females_aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 2 + # Aa × AA ⟶ AA : Aa
Rez["Females_Aa", t, i] * Rez["Males_Aa", t, i] / be_N_Ma / 4) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
} # Закриття дужок підрахунку самиць та самців після else
be_N_Hp <- Rez["Hermaph_AA", t, i] + Rez["Hermaph_Aa", t, i] + Rez["Hermaph_aa", t, i] # Загальна кількість гермафродитів
if(be_N_Hp<2) { # Якщо гермафродитів менше за два, бета-чисельність дорівнює альфа-чисельності
be_N_Hp_AA <- Rez["Hermaph_AA", t, i]
be_N_Hp_Aa <- Rez["Hermaph_Aa", t, i]
be_N_Hp_aa <- Rez["Hermaph_aa", t, i]
} else { # Розраховується (з окремими позначеннями) чисельність різних за складом пар гермафродитів
if(Rez["Hermaph_AA", t, i]>1) Pairs_Herms_AA_AA <- Rez["Hermaph_AA", t, i]*(Rez["Hermaph_AA", t, i]-1)/be_N_Hp else Pairs_Herms_AA_AA <- 0
if(Rez["Hermaph_Aa", t, i]>1) Pairs_Herms_Aa_Aa <- Rez["Hermaph_Aa", t, i]*(Rez["Hermaph_Aa", t, i]-1)/be_N_Hp else Pairs_Herms_Aa_Aa <- 0
if(Rez["Hermaph_aa", t, i]>1) Pairs_Herms_aa_aa <- Rez["Hermaph_aa", t, i]*(Rez["Hermaph_aa", t, i]-1)/be_N_Hp else Pairs_Herms_aa_aa <- 0
Pairs_Herms_AA_Aa <- Rez["Hermaph_AA", t, i]*Rez["Hermaph_Aa", t, i]/be_N_Hp
Pairs_Herms_AA_aa <- Rez["Hermaph_AA", t, i]*Rez["Hermaph_aa", t, i]/be_N_Hp
Pairs_Herms_Aa_aa <- Rez["Hermaph_Aa", t, i]*Rez["Hermaph_aa", t, i]/be_N_Hp
# А тепер - бета-чисельність різних генотипів залежно від кількості можливих схрещувань
be_N_Hp_AA <- Rez["Hermaph_AA", t, i] + floor(hp_O_HpHp *
(Pairs_Herms_AA_AA*2 + # AA × AA ⟶ AA
Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_Aa_Aa/2) + runif(1)) # Aa × Aa ⟶ AA : 2 Aa : aa
be_N_Hp_Aa <- Rez["Hermaph_Aa", t, i] + floor(hp_O_HpHp *
(Pairs_Herms_AA_Aa + # AA × Aa ⟶ AA : Aa
Pairs_Herms_AA_aa*2 + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_Aa_aa) + runif(1)) # Aa × aa ⟶ Aa : aa
be_N_Hp_aa <- Rez["Hermaph_aa", t, i] + floor(hp_O_HpHp *
(Pairs_Herms_Aa_aa + # AA × aa ⟶ Aa
Pairs_Herms_Aa_Aa/2 + # Aa × Aa ⟶ AA : 2 Aa : aa
Pairs_Herms_aa_aa*2) + runif(1)) # aa × aa ⟶ aa
} # Закриття дужок підрахунку гермафродитів після else
be_N_Pg_AA <- Rez["Parthen_AA", t, i] + floor(pg_O_Pg * Rez["Parthen_AA", t, i] + runif(1)) # ...і партеногенетики
be_N_Pg_Aa <- Rez["Parthen_Aa", t, i] + floor(pg_O_Pg * Rez["Parthen_Aa", t, i] + runif(1))
be_N_Pg_aa <- Rez["Parthen_aa", t, i] + floor(pg_O_Pg * Rez["Parthen_aa", t, i] + runif(1))
# III.3.e. Визначення добору для гомозигот та гетерозигот: залишок від поділу визначає, який йде добір
if(t%%2==1) { # Якщо покоління непарне, то...
v_homozygotes <- 1-s # ...чисельність гомозигот скорочується, ...
v_heterozygotes <- 1 # ...а чисельність гетерозигот лишається постійною
} else { # Якщо покоління парне, то...
v_homozygotes <- 1 # ...чисельність гомозигот лишається постійною...
v_heterozygotes <- 1-s} # ...а чисельність гетерозигот скорочується
# III.3.f. Вплив добору на кожну з груп (розрахунок гама-чисельностей)
ga_N_Fe_AA <- floor(be_N_Fe_AA * v_homozygotes + runif(1)) # Додаток кожної бета-чисельності та...
ga_N_Fe_Aa <- floor(be_N_Fe_Aa * v_heterozygotes + runif(1)) # ...життездатності у даному поколінні...
ga_N_Fe_aa <- floor(be_N_Fe_aa * v_homozygotes + runif(1)) # ...зазнає ймовірнісного округлення:
ga_N_Ma_AA <- floor(be_N_Ma_AA * v_homozygotes + runif(1)) # ...це і є гама-чисельність...
ga_N_Ma_Aa <- floor(be_N_Ma_Aa * v_heterozygotes + runif(1)) # ...що розраховується...
ga_N_Ma_aa <- floor(be_N_Ma_aa * v_homozygotes + runif(1)) # ...послідовно для усіх форм
ga_N_Hp_AA <- floor(be_N_Hp_AA * v_homozygotes + runif(1))
ga_N_Hp_Aa <- floor(be_N_Hp_Aa * v_heterozygotes + runif(1))
ga_N_Hp_aa <- floor(be_N_Hp_aa * v_homozygotes + runif(1))
ga_N_Pg_AA <- floor(be_N_Pg_AA * v_homozygotes + runif(1))
ga_N_Pg_Aa <- floor(be_N_Pg_Aa * v_heterozygotes + runif(1))
ga_N_Pg_aa <- floor(be_N_Pg_aa * v_homozygotes + runif(1))
# III.3.g. Створення вектору з гама-складом популяції
Ga_Pop <- c(rep(1.11, ga_N_Fe_AA), rep(1.12, ga_N_Fe_Aa), rep(1.22, ga_N_Fe_aa), rep(2.11, ga_N_Ma_AA), rep(2.12, ga_N_Ma_Aa), rep(2.22, ga_N_Ma_aa), rep(3.11, ga_N_Hp_AA), rep(3.12, ga_N_Hp_Aa), rep(3.22, ga_N_Hp_aa), rep(4.11, ga_N_Pg_AA), rep(4.12, ga_N_Pg_Aa), rep(4.22, ga_N_Pg_aa))
# III.3.h. Створення вектора долі (Fate) і вектора жереба (Lot)
if(length(Ga_Pop)>K) Fate <- rep(NA, length(Ga_Pop)) else Fate <- rep(NA, K) # Визначається довжина вектора долі
Fate[1:K] <- 1 # В векторі долі стільки одиниць, скільки має вижити особин, але вони розташовані впорядковано
Lot <- sample(Fate, length(Ga_Pop), replace = FALSE) # В векторі жереба одиниці та NA випадково переплутані
# III.3.i. Вектор жереба визначає остаточний склад в циклі
Om_Pop <- Ga_Pop * Lot # У векторі Om_Pop лишається K особин; загиблі особини - NA
# III.3.j. Закінчення основного робочого циклу моделі і збереження результатів останнього циклу
FM <- length(which(Om_Pop<3)) # Перевірка кількості трьох форм відтворення...
Hp <- length(which(Om_Pop<4))-FM #...щоб у разі, коли залишається тільки одна...
Pg <- length(which(Om_Pop<5))-Hp # ...перервати цикл та зекономити час
if(FM==0 & Hp==0 | FM==0 & Pg==0 | Hp==0 & Pg==0) break # умова переривання даної ітерації
} # Основний робочий цикл моделі завершився
Rez["Females_AA", cycles+1, i] <- length(which(Om_Pop==1.11)) # Заповнення останнього стовпчику для результатів даної ітерації
Rez["Females_Aa", cycles+1, i] <- length(which(Om_Pop==1.12))
Rez["Females_aa", cycles+1, i] <- length(which(Om_Pop==1.22))
Rez["Males_AA", cycles+1, i] <- length(which(Om_Pop==2.11))
Rez["Males_Aa", cycles+1, i] <- length(which(Om_Pop==2.12))
Rez["Males_aa", cycles+1, i] <- length(which(Om_Pop==2.22))
Rez["Hermaph_AA", cycles+1, i] <- length(which(Om_Pop==3.11))
Rez["Hermaph_Aa", cycles+1, i] <- length(which(Om_Pop==3.12))
Rez["Hermaph_aa", cycles+1, i] <- length(which(Om_Pop==3.22))
Rez["Parthen_AA", cycles+1, i] <- length(which(Om_Pop==4.11))
Rez["Parthen_Aa", cycles+1, i] <- length(which(Om_Pop==4.12))
Rez["Parthen_aa", cycles+1, i] <- length(which(Om_Pop==4.22))
# III.4. Ending of higher level cycles — завершення циклів вищого рівня
} # Кінець циклу ітерацій
Outcome[a, b, "Females+Males"] <- sum(Rez[1:6, cycles+1, ])/(K*iterat)
Outcome[a, b, "Hermaphrodites"] <- sum(Rez[7:9, cycles+1, ])/(K*iterat)
Outcome[a, b, "Parthenogenetics"] <- sum(Rez[10:12, cycles+1, ])/(K*iterat)
} # Кінець циклу, що змінює силу добору
} # Кінець циклу, що змінює значення параметру Мальтуса
## [1] "1 from 27"
## [1] ...
# IV. FINISHING — ЗАВЕРШЕННЯ:
# IV.3. Results viewing — перегляд підсумків
Outcome
## ...
# Results saving — збереження об’єктів з результатами
save(Outcome, file = "Outcome.RData")
# IV.4. Results visualization — візуалізація підсумків
persp3D(z = Outcome[, , "Females+Males"], x = vector_r, y = vector_s, theta = 330, phi = 30, # Побудова графіку
xlab = "\nr\n(плодючість)", ylab = "\n\ns\n(коефіцієнт добору)", zlab = "\n\nPart of females & males\nvictories",
expand = 0.8, facets = FALSE, scale = TRUE, ticktype = "detailed",nticks = 6, cex.axis=0.9,
clab = "Part of\nfemales & males\nvictories", colkey = list(side = 2, length = 0.5))